화학구조 그리기 및 구조형태찾기(conformational search)
1.1 화학구조 그리기 및 에너지 최소화 과정, 용매효과 과정
본 과정의 목적은 CAChe에서 화학구조를 그리고 분자의 낮은 에너지 상태를 찾는 과정, 그리고 용매화 시키는 과정에 대하여 설명하고자 합니다.
1.1.1 화학구조 그리기
먼저 CAChe를 실행시키기 위해서는 시작메뉴의 프로그램에서 CAChe라는 폴더에 workspace를 실행시키면 다음과 같은 윈도우가 나타나게 됩니다.
그러면 좌측에 도구막대(toolbar)가 나오게 되는데 selection tool, drawing tool, manipulation tool로 3가지로 구성되어 있고 상단에는 메뉴(Menu)와 기능관련 도구막대 그리고 리본(ribbon)이 있습니다.
선택도구(selection tool)
![]() 원자, 결합을 선택할 때 사용하는 버튼입니다.
원자, 결합을 선택할 때 사용하는 버튼입니다.
![]() 분자를 선택할 때 사용하는 버튼입니다.
분자를 선택할 때 사용하는 버튼입니다.
![]() 미리 선택된 부분과 유사한 부분(fragment)를 모두 선택할 때 사용하는 버튼입니다.
미리 선택된 부분과 유사한 부분(fragment)를 모두 선택할 때 사용하는 버튼입니다.
그리기도구(drawing tool)
![]() 분자를 그릴 때 사용하는 버튼입니다.
분자를 그릴 때 사용하는 버튼입니다.
조정도구(manipulation tool)
![]() 평면상, 공간상 에서 회전을 시킬 때 사용하는 버튼입니다.
평면상, 공간상 에서 회전을 시킬 때 사용하는 버튼입니다.
![]() 평면상에서 이동할 때 사용하는 버튼입니다.
평면상에서 이동할 때 사용하는 버튼입니다.
![]() 확대 또는 축소를 할 때 사용하는 버튼입니다.
확대 또는 축소를 할 때 사용하는 버튼입니다.
기능관련 도구막대
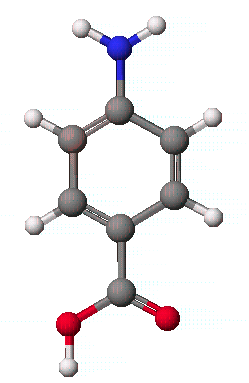
먼저 예로 ρ-aminobenzoic acid(PABA)의 화학구조를 그려보도록 합시다.
ρ-aminobenzoic acid(PABA)는 적혈구 형성에 도움을 주며, 머리카락 색을 자연색을 바꾸어 주는 역할을 하고, 공업적으로는 태양선 차단제로 사용되고 있습니다. ρ-aminobenzoic acid(PABA)가 생체내에서 부족할 경우 극도의 피곤과 습진, 두통과 우울증등을 유발하며 조기에 머리카락색이 희게 되는 현상을 일으키기도 합니다.
먼저 그리기 도구를 이용하여 분자를 그릴 때 원자의 종류와 결합종류 그리고 전하량과 혼성화(hybridization)등을 확인하여야 합니다. 일반적으로 구조의 골격을 모두 탄소로 그린후 선택도구를 이용하여 원자와 결합종류, 전하량, 혼성화 등을 바꾸어 주는 것이 사용하기에 용이합니다.
그리기 도구인![]() 를 선택한 후 메뉴아래에 나타나는 리본(ribbon)에서 원자의 종류는 C로, 혼성은 sp3 tetrahedron으로, 전하는 0으로, 결합종류는 single로 조정합니다.
를 선택한 후 메뉴아래에 나타나는 리본(ribbon)에서 원자의 종류는 C로, 혼성은 sp3 tetrahedron으로, 전하는 0으로, 결합종류는 single로 조정합니다.
원하는 지점에서 마우스의 왼쪽버튼을 한번 누르면 탄소가 하나 생기고, 다시 마우스의 왼쪽버튼을 그 지점에 놓고 누른 상태로 끌어당기면 결합이 생기고, 그렇게 하여 육각형의 링을 만듭니다. 이 상태에서 프로그램 상단에 있는 메뉴에서 Beautify의 Rings를 선택하면, Cyclohexane을 만들 수 있습니다.

그리기 도구를 선택하여 수소를 제외한 나머지 부분들(amino 와 carboxy group)을 Carbon으로 그립니다.
선택도구인 ![]() 를 선택한 후, 해당하는 원자를 선택한 후에 리본에서 원자의 종류를 각각 Nitrogen과 Oxygen으로 바꿉니다. 이중결합으로 만들어야 하는 부분은 결합의 중간부분을 선택하여 리본에서 결합종류를 Single에서 Double로 바꿉니다.
를 선택한 후, 해당하는 원자를 선택한 후에 리본에서 원자의 종류를 각각 Nitrogen과 Oxygen으로 바꿉니다. 이중결합으로 만들어야 하는 부분은 결합의 중간부분을 선택하여 리본에서 결합종류를 Single에서 Double로 바꿉니다.
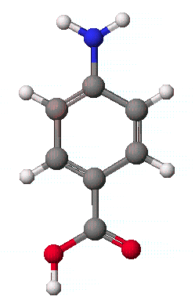
기본적으로 화학구조가 그려지면, Beautify를 이용하여 알맞은 구조를 나타내도록 합니다. 그 종류는 원자가(valence)와 혼성화(hybridization), 링(ring), 기하학적 구조(Geometry)등으로 구분되는데 일반적으로 맨 위에 있는 Comprehensive는 4가지의 기능을 동시에 수행합니다. 기본적인 구조가 그려진 후 Beautify의 Comprehensive를 실행하면 4가지를 모두 만족하는 ρ-aminobenzoic acid의 구조를 얻게 됩니다.
1.1.2 화학구조 열기와 저장하기
열기(Open)와 저장(Save)은 File 메뉴에 있으며, CACHe가 설치되어 있는 폴더 밑에 있는 User라는 폴더가 기본적으로 나타나게 되어 있습니다. 열기와 저장할 때 지원되는 파일 형식은 다음과 같습니다.
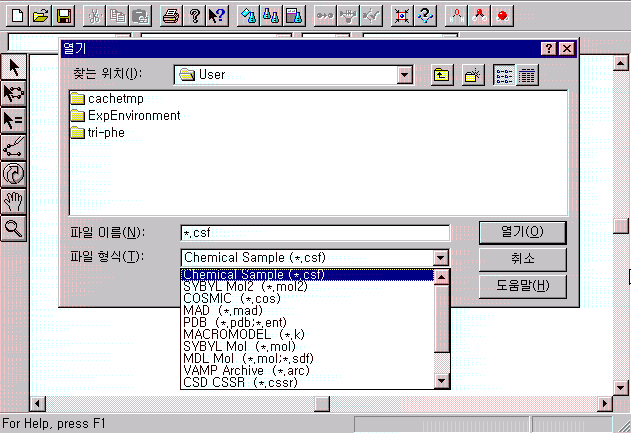
열기(Open) 파일 형식 |
*.csf (CAChe Chemical sample file) : CAChe의 기본적인 화학적 구조 파일 형식 *.mol2 (Sybyl mol2 file) : SGI용 Tripos사의 Sybyl 프로그램의 파일 형식 *.cos (COSMIC file) *.mad (MAD file) *.pdb, *.ent (Protein Data Bank file) : Protein Data Bank로 지원되는 파일 형식 *.k (MACROMODEL file) : MACROMODEL의 파일 형식 *.mol (Syble mol file) : SGI용 Tripos사의 Sybyl 프로그램의 파일 형식 *.mol (MDL mol file) : MDL사 프로그램의 mol 파일 형식 *.arc (VAMP file) : Oxford사의 VAMP 프로그램의 파일 형식 *.cssr (CSD CSSR file) *.map (CAChe Map file) : CAChe에 의해 계산된 energy map 파일 형식 *.exp (CAChe Experiment file) : CAChe에서 실험내용을 저장한 파일 형식 |
| 저장(Save) 파일 형식 |
*.csf (CAChe Chemical sample file) : CAChe의 기본적인 화학적 구조 파일 형식 *.mol2 (Sybyl mol2 file) : SGI용 Tripos사의 Sybyl 프로그램의 파일 형식 *.pdb, *.ent (Protein Data Bank file) : Protein Data Bank로 지원되는 파일 형식 *.mol (Syble mol file) : SGI용 Tripos사의 Sybyl 프로그램의 파일 형식 *.mol (MDL mol file) : MDL사 프로그램의 mol 파일 형식 *.smi (SMILES string file) *.cssr (CSD CSSR file) |
그 밖에 Internet에서 무료로 공급되는 Babel이라는 프로그램을 통하여 다양한 분자모델링 프로그램 파일 형식으로 전환이 가능합니다. 특히 CAChe의 csf 파일은 양자계산용 Gaussian 프로그램 안에 있는 NewZmat라는 프로그램에 의해 파일전환이 가능하며, Gaussian으로 계산한 결과도 NewZmat를 이용하여 CAChe로 전환이 가능합니다.
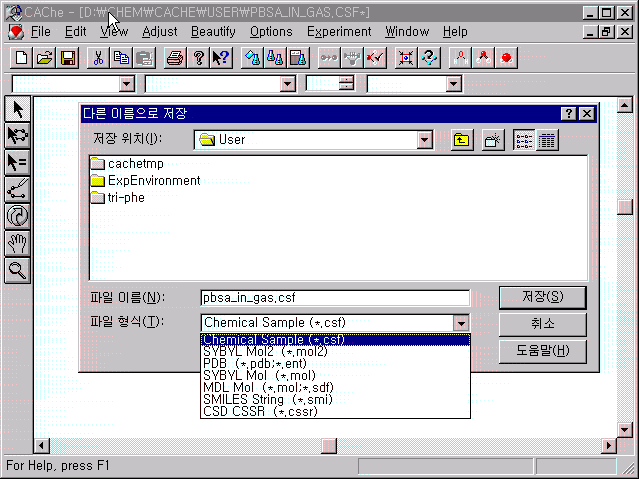
앞 과정에서 ρ-aminobenzoic acid의 구조는 File 메뉴에 있는 Save(저장)로 가서 pbsa_in_gas라는 이름으로, 형식은 Chemical Sample(*.csf)으로 저장하도록 합니다.
1.1.3 기체상에서의 생성열(heat of formation) 계산
메뉴에서 Experiment의 New를 선택하고 다음과 같은 원도우가 나타나게 됩니다.
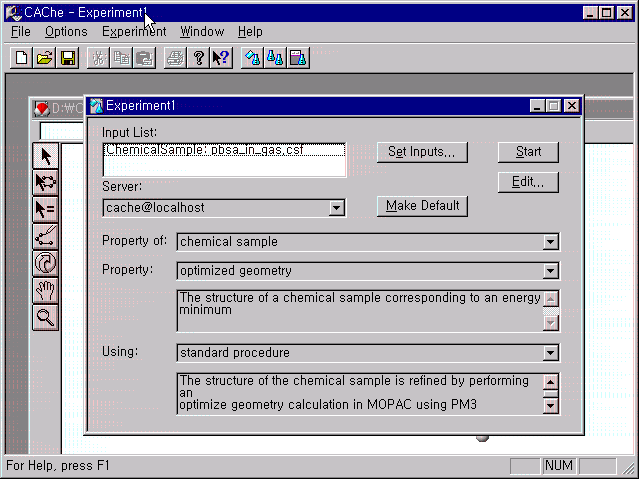
Experiment에 의한 계산 결과는 지정한 파일이름과 같은 이름의 폴더가 생기면서 저장됩니다.
만일 화학구조를 저장을 하지 않고 Experiment를 진행하면 cachetmp라는 곳에 저장되게 됩니다.
input list에는 Experiment를 수행할 파일이름이 나와 있으며, Set input을 이용하여 다른 파일을 불러 실행할 수 있습니다. 또한 CAChe Workstation version의 경우 Server가 설치되어 있으면, 계산을 Server로 수행할 수 있습니다. 일반적으로 CAChe Workstation이 설치되어 있는 컴퓨터에 계산을 수행하도록 기본값으로 제공되며, server에는 cache@localhost로 표시됩니다.
먼저 기체상에서의 생성열(heat of formation)을 계산하기 위하여, 그림에서와 같이
property of : chemical sample로, property : optimized geometry로, Using : MM/AM1 geometry로 3가지를 선택합니다.
property of 의 경우 chemical sample, atom, bond, chemical sample conformation, reaction and transtion states등이 있으며, 그려진 화학구조에의 특성을 조사할 경우는 chemical sample로, 각 atom의 부분 전하를 조사할 경우에는 atom으로, bond order나 bond strain energy를 조사할 경우는 bond를 선택합니다. 그리고 형태 찾기(Conformational search)를 할 경우는 chemical sample conformation를 선택하고, 반응이나 전이상태(transition state)의 구조를 찾을 경우에는 reaction and transtion states를 선택하도록 합니다.
property 의 경우는 property of에 따라 수행할 수 있는 특성들이 각각 달라지게 됩니다.
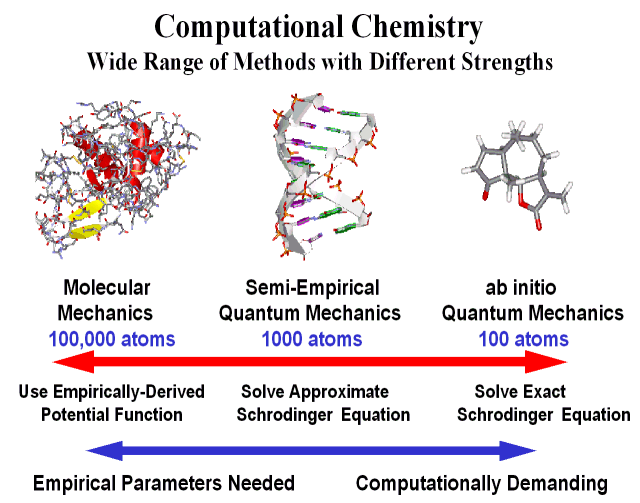
Using은 계산방법을 결정하는 것으로 일반적으로 모든 분자모델링용 프로그램에서 지원하는 계산 방법은 크게 Molecular Mechanics, Semi-empirical method, Ab initio method등 3가지로 구분할 수 있습니다. 간단히 설명하면 다음과 같습니다.
Molecular Mechanics : 원자를 기본단위로 근사를 배제하고 계산에 필요한 결합길이, 결합각도, 비틀림각 등을 기존의 실험에서 얻은 값들을 이용하여 분자의 안정한 구조를 결정하는 방법입니다. 다른 방법에 비해 계산속도가 빠르고 생체분자등과 같이 매우 큰 분자도 계산이 가능합니다. 기본적으로 Molecular Mechanics방법에는 다양한 Force Field들이 있는데, 각각의 Force Field에는 핵의 위치에 알맞은 부분 전하를 계산하기 전에 미리 설정하여 계산하며, 전자적인 효과를 볼 수는 없습니다. CAChe에서는 MM2와 MM3가 이에 해당합니다. Semi-empirical method : 양자역학(Quantum mechanics)방법에 기반을 한 방법으로, 원자가전자만을 고려하고, 내부전자들은 핵에 의하여 차폐되어 있다고 가정한 방법입니다. 그러므로 계산값이 실험값과 일치하도록 많은 시간이 요하는 부분을 실험적으로 얻은 매개변수를 사용하며, 수백개정도의 원자를 가지고 있는 분자들을 계산할 수 있습니다. 계산방법에 따라 해당하는 원자의 매개변수가 있어야 하므로 계산하기 전에 해당원자의 매개변수를 가지고 있는지 확인하여야 하며, 전자분포에 대한 정보를 얻을 수 있습니다. CAChe에서는 MOPAC이라는 프로그램이 내장이 되어 있으며 AM1과 PM3와 같은 semi-empirical method들을 가지고 있습니다. 다소 차이는 있지만, 이것은 3주기 원자들까지 사용할 수 있습니다. 4주기의 전이금속의 경우 ZINDO가 지원 가능합니다. 원자의 매개변수 지원여부를 확인하려면 CAChe manual을 참고하세요. Ab initio method : 실험결과에 의존함 없이 순수한 이론적인 방법으로 원자내의 모든 전자들을 고려하여 분자궤도함수를 계산하는 방법입니다. 많은 계산시간을 필요로 하며 일반적으로 수십개의 원자로 구성된 분자들까지 계산이 가능합니다. 이방법중에서 CAChe는 DFT(density functional theory)에 입각한 DGauss라는 프로그램을 CAChe의 부속모듈로 판매하고 있습니다. |
계산 방법들을 간단히 정리한 그림은 다음과 같습니다.
Using에서 MM/AM1 geometry를 선택하면, 시간이 적게 소요되는 MM3 방법으로 먼저 안정한 구조를 결정한 후에 최종적으로 Semi-empirical방법인 AM1방법을 사용하여 구조를 최적화 시킵니다.
Experiment의 설정이 다 되면, Start를 눌러서 계산을 수행하고 다음과 같은 원도우가 계산 수행과정에 관한 정보를 제공하게 됩니다.
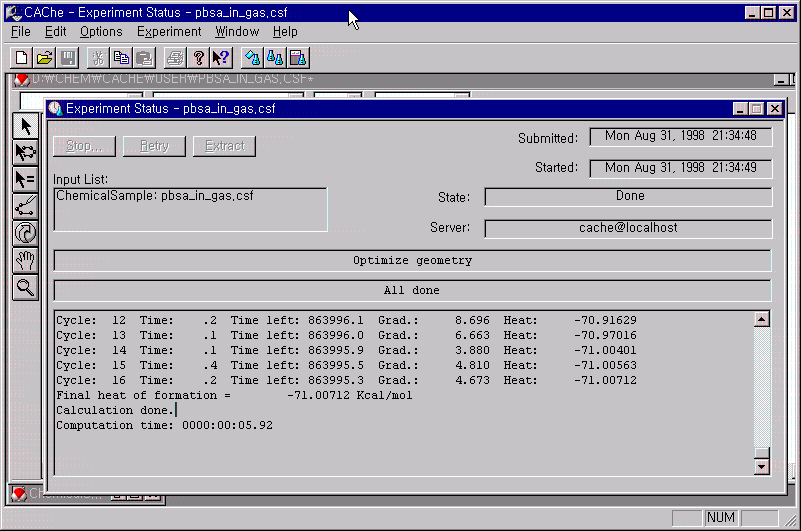
MOPAC 계산이 수행중일 경우 state는 pending으로 표시되며, 종료되면 Done으로 표시되고 그 결과에 따라서 분자의 구조가 자동으로 변화합니다.
따라서 ρ-aminobenzoic acid구조가 그려진 원도우를 선택하면, MOPAC결과에 의해 최적화된 분자구조를 확인할 수 있고, 최적화된 구조가 다시 파일로 저장되지 않았으므로 파일명뒤에 *가 붙어 있습니다. 이것은 저장을 하게 되면 *가 없어지게 됩니다.
또한 파일이름과 같은 PASA_in_gas.io라는 이름의 폴더가 생성되고, 그안에 log 파일과 out 파일이 생성됩니다. 자세한 계산결과는 Molecular Mechanics의 경우 mechancs.out에, AM1결과는 mopac.out를 참조하길 바랍니다.
ρ-aminobenzoic acid의 기체상에서의 생성열(Heat of formation)값은 결과의 아래에 보이는 것과 같이 -71.00712 Kcal/mol에 해당됩니다.
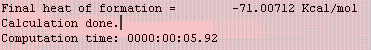
1.1.4 수용액상태에서의 생성열(heat of formation) 계산
기체상에서의 최적화된 구조를 이용하여 수용액상태의 생성열을 계산하도록 하겠습니다.
먼저 기체상에서 최적화된 구조가 그려진 원도우로 이동한 후에, Edit 메뉴안에 있는 Copy를 선택하여 구조를 복사하도록 합니다. 그 다음 File 메뉴안의 New를 선택하여 새로운 원도우를 열고, Edit 메뉴의 paste를 선택하여 복사한 구조를 붙이도록 합니다.
새로운 원도우를 저장하도록 하며, 그 이름을 PABA_in_water로 합니다.
다시 Experiment 메뉴안의 New를 선택하고 아래와 같은 창이 나타나면 Using을 MM/AM1 geometry in water로 선택하도록 합니다.
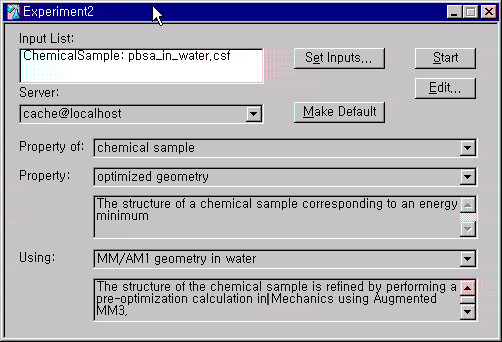
water에서의 계산은 MOPAC 프로그램의 COSMO(Conductor-like Screening Model)이라는 모델을 이용하여 계산하는데, 이것은 원자의 van der Waals반지름과 solvent의 effective radius를 기반으로한 polygonal한 전도성 표면을 만들고, 그 표면의 전하상(image of charge)에 의해 용매의 유전 차폐 에너지(dielectric screening energy)를 근사한 방법입니다. 특히 수용액의 경우, 좋은 근사법으로 알려져 있으며, 이 계산을 수행할 경우, 용매가 접근할 수 있는 표면적(solvent accessible area)를 얻을 수 있습니다.
계산이 완료되면, ρ-aminobenzoic acid의 수용액에서의 생성열(Heat of formation)값은 결과의 아래에 보이는 것과 같이 -89.08384 Kcal/mol에 해당됩니다.
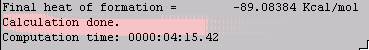
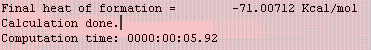
기체상과 수용액상에서의 결과를 비교하였을 때, 에너지 측면에서는 ρ-aminobenzoic acid는 용해되는 것이 안정하지만, 실제로 용해성에 관련된 정보는 그밖에 다른 상호작용도 고려하여야 합니다.
Water heat of formation | – | Gas phase heat of formation | = | Heat of Solvation |
-89.08384 Kcal/mol |
| -71.00712 Kcal/mol |
| -18.07672 Kcal/mol |
1.2 형태 찾기(Conformational search)
1.2.1 배경
어떠한 분자모델링 프로그램으로 그려진 화학구조를 최적화하면 국부 최소점(local minimum)으로 이동하며 언제나 전체 최소점(global minimum)으로 이동하는 것은 아닙니다. 그러므로 형태찾기(conformational search)는 가장 낮은 에너지상태의 구조를 찾는 데에 필요한 과정입니다. 일부는 에너지 장벽 너머로 존재하는 국부 최소점들이 화학적으로 관심에 대상이 되기도 합니다.
1.2.2 분자구조 그리기
본 교재에서는 이러한 형태찾기의 예로 3-amino-propaldehyde를 사용하도록 하겠습니다.
3-amino-propaldehyde
먼저 5개의 탄소를 그린 후 ![]() 를 이용하여 산소와 질소부분으로 바꾸어 주도록 합니다.
를 이용하여 산소와 질소부분으로 바꾸어 주도록 합니다.
기본 골격구조가 그려지면 Beautify 메뉴에서 Comprehensive를 선택하여 위와 같이 적당한 구조형태를 같도록 합니다. 이 과정이 익숙하지 않으시면 1.1.1과정으로 가서 다시 보시기 바랍니다.
1.2.3. 형태찾기 라벨(Conformational search label) 결정
모든 가능한 형태의 구조를 찾기 위해서는 먼저 2개의 탄소-탄소 결합을 회전시킬때의 에너지 변화를 알아볼 필요가 있습니다. 그러려면 회전시킬 2개의 이면각(dihedral angle)를 정의하여야 합니다.
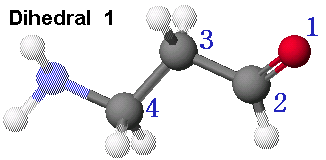
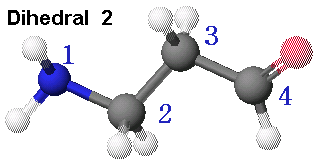
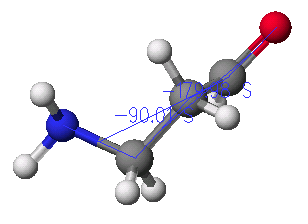
그림에서와 같이 먼저 첫 번째 이면각(dihedral angle)은 산소를 먼저 선택하고, 그 다음 가까운 탄소 순으로 4개의 원자를 선택합니다. 이때 여러 개의 원자를 선택할 때는 자판에서 shift 키를 누른 상태에서 선택하여야 합니다. 또한 선택할 때의 순서도 중요한데, 이것은 1번 원자가 2-3-4원자를 기준으로 하여 회전을 하기 때문입니다. 따라서 선택도구를 이용하여 원자들을 선택할 때도 최종적으로 결과를 얻어 낼 때를 고려하여 순서를 결정하여야 합니다.
선택하고 난후, Adjust 메뉴에 있는 Dihedral angle를 선택하면 다음과 같은 원도우가 나오게 됩니다.

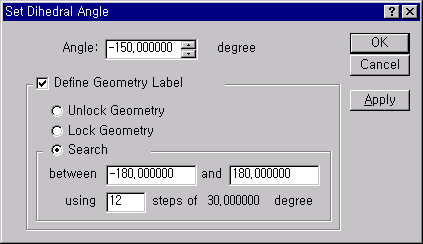
이 때 현재의 각이 Angle에 나타나게 되며, Search를 선택하여 회전할 범위와 단계(step)수를 결정하도록 합니다. 일반적으로 단일 결합의 경우는 30 또는 60도를 한 단계(step) 범위로 설정합니다.
위와 같은 과정으로 두 번째 이면각(dihedral angle)도 정의해 주도록 합니다.
형태찾기 라벨(conformational search label)를 결정하는 단계가 끝나면 위의 과정을 확인하는 과정이 필요합니다. Adjust 메뉴에서 Animate Geometry Labels를 선택하면 아래와 같은 원도우가 열기고 play를 시켜서 회전하는 부분이 정확하게 정의되었는지를 확인하도록 합니다.
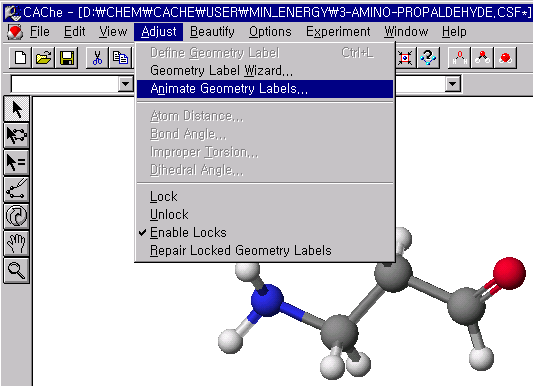
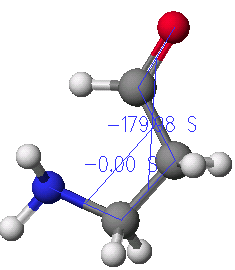
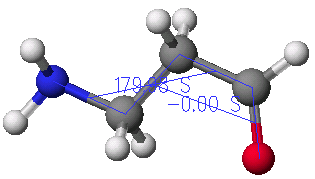

다소 화합물이 커서 회전시킬 이면각이 많을 경우에는 회전할 범위와 단계(step)수를 경험적으로 적게 설정할 경우, 계산할 양이 크게 적어지게 됩니다. 예를 들어 10개의 이면각을 지니고 있는 분자의 경우 60도씩 6번 회전시킬 경우 610 = 60,466,176 번의 계산을 수행하여야 합니다. 그러나 구조 최적화에 대한 경험으로 5개의 이면각은 90도씩 회전시켜도 될 경우에는 45×65 = 7,962,624 번의 계산을 수행하게 되므로 무려 8분의 1정도로 계산량을 줄일 수가 있습니다.
이면각이 4개 미만인 경우에는 아래의 그림과 같이 Adjust 메뉴 안의 Geometry Label Wizard를 선택하면, 단일결합과 이중결합 그리고 약한 결합에 대한 이면각의 회전을 자동으로 설정할 수 있도록 되어 있습니다. 보통 terminal group에 대해서는 이면각을 조사하지 않는데, 그 부분의 이면각도 포함시키려면 include terminal groups를 선택하여야 합니다.
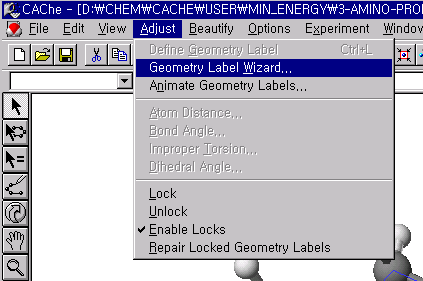
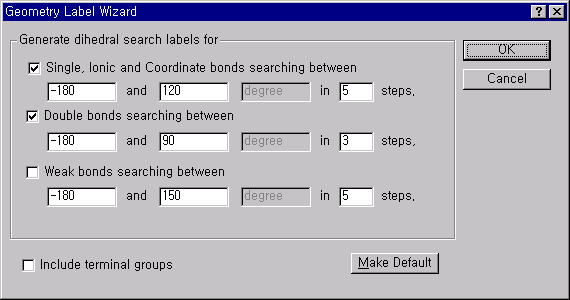
모두 정확하게 설정되었으면, 분자구조를 저장하고 Experiment 과정으로 들어가도록 합니다.
1.2.4. 형태찾기(Conformational search label) 계산 과정
Experiment 메뉴안의 New를 선택하면 아래와 같은 원도우가 열리게 되고, Property of 는 chemical sample conformations로, Property는 Optimzed map으로, Using은 fastest procedure로 선택합니다.
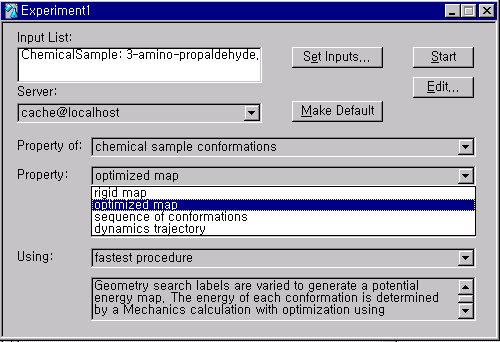
Chemical sample conformations은 다양한 형태찾기(conformational search)과정을 수행할 때 선택하는 것입니다. Property의 경우는 rigid map, optimized map, sequence of conformations, dynamics trajectory로 구성되어 있습니다.
Rigid map의 경우는 주로 단계수를 많이 하였을 때 사용하는 것으로 다양한 형태의 구조를 최적화 시키지 않고 바로 그 지점(single point calculation)의 에너지 상태를 알아보는 것입니다. 특히 계산하는 사람이 흥미를 갖는 부분만을 라벨로 지정하여 에너지 변화를 볼 경우에 유용한 방법입니다. 이 경우에는 Molecular Mechanics방법을 이용할 경우 최대 8개까지의 라벨을 설정할 수 있고, Quantum Mechanics방법을 이용할 때는 최대 2개까지의 라벨을 지정할 수 있습니다.
Optimized map은 다양한 형태의 구조를 에너지적으로 최적화하는 과정을 수행하도록 하는 것이며, Molecular Mechanics방법을 이용할 경우 최대 4개까지의 라벨을 설정할 수 있고, Quantum Mechanics방법을 이용할 때는 최대 2개까지의 라벨을 지정할 수 있습니다.
Sequence of conformations은 형태찾기 라벨(conformational search label)의 제한 없이 사용할 수 있으며, 각각의 단계를 일렬로 연결한 형태로 나타냅니다. 이 경우는 Molecular Mechanics방법만 사용할 수 있습니다. 아래의 그림과 같이 sequence of conformations를 선택하면 Using의 경우 one pass 와 multiple pass로 나누어 지는데, one pass의 경우는 각각의 라벨이 독립적으로 계산되는 것을 말합니다. 즉 한가지 라벨을 변화시켜 에너지적으로 낮은 구조를 찾으면, 그 라벨을 고정시킨 뒤 다음 라벨을 변화시켜서 낮은 구조를 찾는 과정을 말합니다. Multiple pass는 모든 영역을 다 찾는 경우에 해당합니다. 예를 들어 회전시킬 이면각이 5개가 있고 60도씩 6번회전시킬 경우 one pass방법으로 수행하면 6 + 6 + 6 + 6 + 6 = 30번을 수행하지만, multiple pass로 수행할 경우에는 65 = 7776번을 수행하게 됩니다. one pass의 경우는 많은 범위를 제외하므로 전체 최소점(global minimum)을 제외시킬 문제점이 있지만 빠르게 계산결과를 얻을 수 있고, multiple pass의 경우에는 전제 범위를 모두 찾으므로 반드시 전체최소점(global minimum)을 찾을 수 있으나, 상당한 계산시간을 필요로 하게 됩니다.
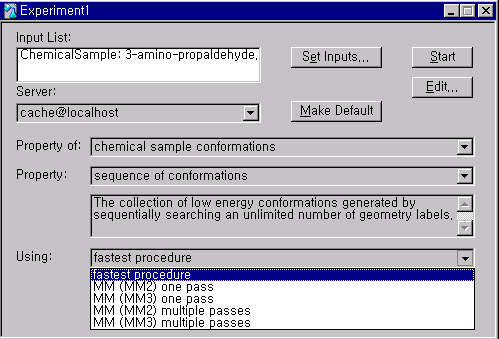
Dynamics trajectory는 절대온도 300K에서 분자의 random한 운동을 볼 수 있고, 결정한 라벨의 변화를 나타냅니다.
계산 단계의 설명이 너무 길었던 것 같습니다. 더 자세한 내용은 CAChe manual를 참고하시길 바랍니다. 그럼 처음으로 돌아가서 결정된 조건에서 Start부분을 누르면, 계산이 수행되고 종료되면 다음과 같은 두 개의 원도우이 나타나게 됩니다.

계산 종료와 더불어 파일 이름과 동일하고 끝에 .io가 붙은 폴더가 생성되는데 이것은 앞부분에서 설명한것과 같이 계산과정을 나타내는 log파일과 계산결과를 나타내는 out파일이 들어 있습니다. 또한 파일이름과 동일하고 끝에 .map이라고 써있는 폴더가 생기게 되는데 이것은 3D 원도우에 나타난 그래프 파일과 각각의 구조를 포함하는 energy.map이라는 파일이 들어 있습니다.
처음 3D 그래프 원도우가 나타날 때 표면위에 나타나 있는 점은 가장 낮은 에너지 구조의 에너지 점을 나타내며 그때의 구조가 옆에 나타나게 됩니다.
여기서 Edit 메뉴안의 Animate Along Axis를 선택하면 에너지 표면위에서 점이 이동하는 모습을 볼 수 있고, 그때의 구조를 다른 원도우를 통해서 볼 수 있습니다.
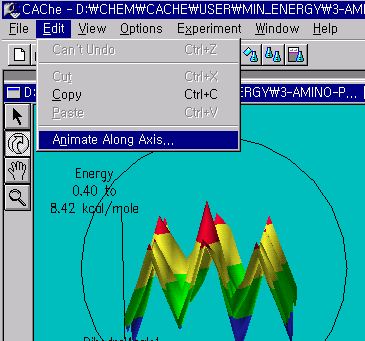
결과적으로 가장 낮은 에너지의 구조는 NH와 CO사이에 intra-hydrogen bond를 형성하는 있는 구조로 나타났으며, 거리는 2.290Å를 나타내었습니다. 실제로 aldehydic O와 amine H의 거리가 1.8∼2.3Å이내이면 수소결합을 하고 있는 증거로 볼 수 있습니다.

